脑肿瘤切除后手术相关失语风险的量化
外科医生在切除脑肿瘤时能否量化失语症的风险?为了找到答案,慕尼黑工业大学 (TUM) Rechts der Isar 诊所的研究人员正在将大脑作为一个网络进行分析。在最近一项针对 60 名患者的研究中,他们的预测达到了四分之三的命中率。慕尼黑工业大学的科学家使用一种特殊形式的磁共振成像(称为纤维束成像)创建大脑神经通路网络的 3D 表示。图片来源:慕尼黑工业大学 (TUM) 脑肿瘤相对罕见。根据德国神经病学协会的数据,每年的发病率约为每 10 万居民 5 例。 “但在大多数情况下……

脑肿瘤切除后手术相关失语风险的量化
外科医生在切除脑肿瘤时能否量化失语症的风险? 为了找到答案,慕尼黑工业大学 (TUM) Rechts der Isar 诊所的研究人员正在将大脑作为一个网络进行分析。 在最近一项针对 60 名患者的研究中,他们的预测达到了四分之三的命中率。
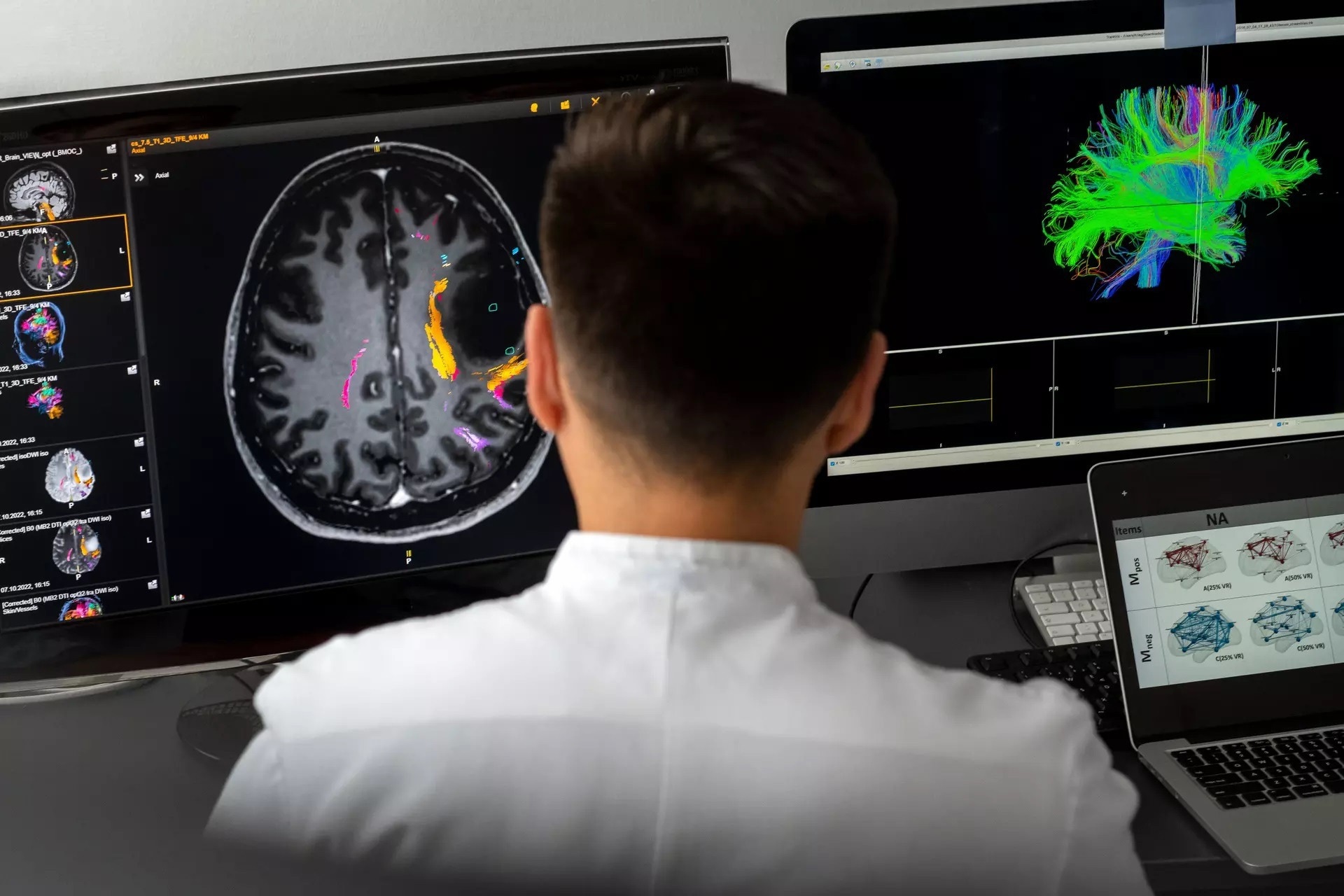
Mit einer speziellen Form der Magnetresonanztomographie, der sogenannten Traktographie, erstellen Wissenschaftler der TUM 3D-Darstellungen der Netzwerke von Nervenbahnen im Gehirn. Bildnachweis: Technische Universität München (TUM)
脑肿瘤相对罕见。 根据德国神经病学协会的数据,每年的发病率约为每 10 万居民 5 例。 “但在大多数情况下,手术切除肿瘤是不可避免的,”Sandro Krieg 教授说,他估计神经胶质瘤(一种常见的脑肿瘤)会在慕尼黑工业大学 (TUM) 的 Klinikum Rechts der Isar 切除。 “几乎每天。”
根据肿瘤的不同,克里格和他的同事制定了个体化治疗和手术策略。 关键一点:应尽可能保护健康组织,并且不应损坏任何可能导致进一步限制的结构。 例如,“失语症”是术后语言障碍的术语。 “我们希望在手术前准确了解失语症的风险。”
Rechts der Isar 诊所神经外科诊所的主任医生十多年来一直从事术前脑部绘图工作。 “我们早就知道大脑中负责运动或语言等功能的中心位置。但直到最近五年,我们才开始分析大脑网络,以了解不同区域如何协同工作,例如,使一个人能够说话。有一点很清楚:不存在这样的语言中心。相反,该结构更像是一个大型网络的几个枢纽或节点,通过它使语言成为可能。”
脑肿瘤:通过机器学习进行预测
对大脑网络特性的分析——所谓的连接组分析——克里格教授的团队已经使用了大约两年的程序——在当前的研究中发挥着关键作用。 “通过这种方式,我们量化了各个大脑区域的连接,”克里格教授说。 “我们现在开始为大脑区域分配更精确的功能。”
神经科学电子书
去年热门采访、文章和新闻的汇编。 下载免费副本
慕尼黑工业大学的科学家张浩苏博士和塞巴斯蒂安伊勒博士绘制了负责语言能力的大脑各层的解剖图像。 该过程如下:“使用特殊形式的磁共振成像、纤维束成像,我们创建大脑神经通路网络和子网络的 3D 表示,”张解释道。
这种网络分析得到了导航经颅磁刺激方法的支持,其中目标磁脉冲抑制负责语言的纤维束中的神经细胞。 这会导致患者出现暂时的言语障碍,可以通过视频分析检测到。 它使研究人员能够精确识别负责语言的大脑区域。
我们将纤维束成像中所谓的连接组参数与患者言语功能的信息结合起来。”
张浩苏博士,慕尼黑工业大学科学家
张和伊勒的算法的特别之处在于,它提供了“具有统计意义的参数”——可用于训练机器学习模型的数据,从而定位个体患者的语言。 尽管不同分析方法的应用看起来很复杂,但该方法的主要特点是简单:整个分析过程不需要复杂的算法或强大的计算机。 “我们使用的数据来自医院的常规测试,”张说。
网络分析:预测语言障碍的准确度为 73%
Rechts der Isar Clinic 的研究人员最近对 60 名患者进行的一项研究表明,这种综合分析可以相当准确地 (73%) 预测手术是否会导致语言困难(术后失语症)。 “能够做出这些预测非常重要,”克里格说。 他对能够通过“真实网络分析”更准确地量化风险并拥有具体数据来帮助绘制大脑图谱的可能性感到兴奋。
更重要的是:在机器学习的帮助下,随着时间的推移,预测会变得更好。 但要做到这一点,研究人员需要更多的患者数据来训练机器学习算法。 “这是唯一可以使用大数据来预测外科手术风险的方法,”克里格教授说,他现在计划招募更多患者进行研究。 他认为,只需“几百”名患者就足以进行高度准确的预测。
来源:
参考:
伊勒,S.,等人。 (2022) 术前功能特异性连接组分析可预测神经胶质瘤切除术后与手术相关的失语症。 绘制人类大脑图。 doi.org/10.1002/hbm.26014 。
。

 Suche
Suche
 Mein Konto
Mein Konto
