Automatisiertes Zebrafisch-Screening zur Entdeckung myeloischer Leukämie-Medikamente

In diesem Interview spricht NewsMedical mit Elspeth Payne und Alexandra Lubin, Krebsforscherinnen am UCL Cancer Institute in London, darüber, wie sie mithilfe von KI-gestützter Analysesoftware mithilfe von Zebrafischmodellen myeloische Leukämien automatisiert mit hohem Durchsatz untersuchen. Athena. Diese erschwingliche Softwarelösung steht zum Download auf einer neuartigen Pay-per-Use-Basis zur Verfügung.
Könnten Sie sich bitte vorstellen, was Sie tun und kurz Ihre Forschung beschreiben?
Elspeth Payne: Mein Name ist Beth Payne und ich bin klinische Wissenschaftlerin. Mein Labor arbeitet an Blutkrebs, insbesondere myeloischen Malignomen, und ich betreue auch Patienten im Krankenhaus. Wir verwenden eine Kombination aus Zebrafisch-Modellierung und Arzneimittelscreenings neben primärem Patientenmaterial, um zu studieren und besser die Entwicklung verstehen myeloische Leukämienund verschiedene Möglichkeiten, wie wir sie in Zukunft behandeln könnten.
Alexandra Lubin: Mein Name ist Alex Lubin. Ich bin Postdoktorand im Labor von Beth Payne. Ich arbeite an Zebrafischmodellen für myelodysplastische Syndrome (MDS) und akute myeloische Leukämie (AML). Arzneimittelscreening ist mein Hauptprojekt, und ich möchte ein Arzneimittelscreening in hämatopoetischen Stammzellen im Schwanz von Zebrafischen durchführen, um nach synthetischer Letalität zu suchen.
Wie haben Sie die Analyse von Zebrafischen durchgeführt, bevor Sie die Athena-Software verwendet haben?
Alexandra Lubin: Wenn ich mir in meinem Medikamentenscreening Stammzellen anschaue, versuche ich, eine Veränderung der Zellzahl in einem Fisch zu messen. Um dies manuell zu tun, müssen alle einzelnen fluoreszierenden Zellen im Schwanz gezählt werden. Das heißt, man muss jeden Fisch unters Mikroskop nehmen, durchgehen und zählen, was sehr zeitaufwändig ist. Aus diesem Grund haben wir begonnen, nach einem automatisierten Ansatz zu suchen.
Vor welchen Herausforderungen stehen Sie, wenn es darum geht, die benötigten Daten zu erhalten?
Beth Payne: Wir haben einige Methoden zum Drogenscreening entwickelt, und obwohl der Zebrafisch ein großartiges Modell dafür ist, war es eine Herausforderung, den richtigen Durchsatz zu erzielen. Obwohl es viele verschiedene Plattformen gibt, die man verwenden kann, haben sie in der Regel maßgeschneiderte Lösungen für die Analyse der Daten. Das bedeutet, dass die Entwicklung jedes neuen Bildschirms sowohl kostspielig ist als auch lange dauert, bis Sie etwas bekommen, das für Ihren Zweck funktioniert.
An welchem Punkt haben Sie sich dann von der manuellen Bildgebung und Bildanalyse entfernt und nach automatisierten Lösungen gesucht?
Beth Payne: In Bezug auf automatisiertes Screening haben wir wahrscheinlich in den letzten fünf bis zehn Jahren nach einer Lösung gesucht. Wir haben uns diese Herausforderung alle paar Jahre als Hauptziel erneut angesehen und uns gefragt: „Wie können wir dies zeiteffizienter gestalten?“ Bildgebendes und manuelles Zählen von Zellen ist für eine ordnungsgemäße Datenquantifizierung erforderlich, aber wir dachten, dass es eine enorme Zeitersparnis wäre, wenn es automatisiert werden könnte. Nicht nur für die Zeit, die für diese Schritte benötigt wird, sondern auch, um eine ganze Reihe von Möglichkeiten zu eröffnen, eine solche Plattform für andere Experimente zu nutzen.
Was sind die größten Herausforderungen beim Sampling mit einem manuellen Workflow?
Alexandra Lubin: Die größte Herausforderung bei der manuellen Methode ist, dass sie zeitaufwändig ist. Es ist nicht besonders schwierig, aber es bedeutet, Stunden um Stunden am Mikroskop zu verbringen. Das andere Hauptproblem dabei ist, dass Sie keine großen Stichprobenzahlen erreichen können. Wenn Sie so etwas wie einen Drogenscreening durchführen möchten, liegt die Stärke in der Fähigkeit, viele Proben zu analysieren.
Was hat diese Technologie Ihrem Labor gebracht?
Beth Payne: Was für dieses System hilfreich war, ist, dass wir es zusätzlich zum Arzneimittelscreening für alle Indikationen verwenden. Wenn wir zum Beispiel ein transgenes oder sogar ein In-situ-Objekt haben, das wir abbilden und im Detail mit hohem Inhalt betrachten möchten, verwenden wir die Athena anstatt es manuell zu tun.
Das andere großartige an dem System ist, dass es flexibel ist und Sie es relativ schnell für einen anderen Assay optimieren können, da das System benutzerfreundlich ist. Das Unternehmen bietet viel Unterstützung, um uns zu helfen, wenn wir auf Schwierigkeiten stoßen.
Ich würde sagen, dass es uns insgesamt ermöglicht, unsere Experimente viel besser anzutreiben, weil wir statt nur 20 Embryonen 200 für die gleiche Verarbeitungszeit verwenden können. Tatsächlich dauert es erheblich weniger Zeit, als es sonst benötigt hätte. Insgesamt bietet es mehr Flexibilität in Bezug auf die Experimente, die wir planen können, und die Art von Leistung, die wir aus der Datenausgabe ziehen können Athena.
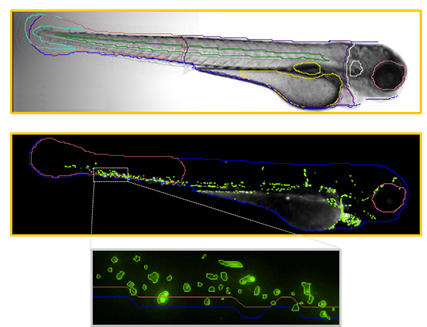
KI-gestützte Analyse eines Hellfeldbilds einer Zebrafischlarve zur Identifizierung der inneren Anatomie in Kombination mit dem Nachweis von GFP-markierten hämatopoetischen Stammzellen. GFP-markierte Zellen, die nur im Schwanz vorhanden sind, können selektiv gezählt werden. Bildnachweis: IDEA Bio-Medical
Azo: Als Sie mit der Arbeit an der Athena-Software begonnen haben, wie lange haben Sie gebraucht, um damit unabhängig und produktiv zu sein?
Alexandra Lubin: Als ich das erste Mal auf die gestoßen bin Athena Software, es war sehr viel in der Entwicklung. Das gab Raum für viele Diskussionen mit IDEA Bio-Medical darüber, was wir von der Software erwarten, was nützlich wäre und Bereiche für Verbesserungen hervorheben.
Nach der Inbetriebnahme war die Übergangszeit von der ersten Nutzung der Software bis zur völligen Unabhängigkeit damit relativ kurz.
Wie sieht das im Vergleich zu anderen Instrumenten aus, die Sie verwenden, zum Beispiel im Vergleich zu manuellen Mikroskopen?
Alexandra Lubin: Dies war insofern eine einzigartige Erfahrung, als wir mehr Kontakt mit IDEA Bio-Medical hatten, als wir es normalerweise tun würden. Ich habe noch nie ein anderes System wie dieses verwendet, daher habe ich keinen direkten Vergleich, aber es ist eine hervorragende und äußerst praktische Software.
Wie lange brauchen Sie, um alle Ihre Fischproben zu analysieren, und wie sieht das im Vergleich zu früheren Zeiten aus?
Alexandra Lubin: Wenn wir diese Experimente manuell durchführen, können wir nur etwa hundert Fische gleichzeitig untersuchen. Das dauert jedoch so viel Zeit, aber sobald wir anfingen, die Athenawir können diese Zahl erhöhen.
Wie viele Fische ich screene, hängt davon ab, wie brav die Fische sind, wie jeder, mit dem man arbeitet Zebrafisch wirst wissen. Es ist möglich, Hunderte (von Fischen) in kurzer Zeit zu screenen, da zwischen dem Beladen der Platte und dem Erhalt der Ergebnisse für etwa hundert Fische nur etwa 30 Minuten vergehen, während dies früher einen ganzen Tag gedauert hätte.
Was tun Sie, wenn Sie Unterstützung bei der Verwendung der Software benötigen? Wer unterstützt Sie und wie?
Alexandra Lubin: Wenn ich Unterstützung bei der Software benötige, wende ich mich an das Team von IDEA Bio-Medical. Sie waren wirklich hilfreich und bieten eine partnerschaftliche Zusammenarbeit an. Ich versorge sie mit Bildern; sie unterstützen uns – es funktioniert reibungslos.
Was war der Punkt, an dem Sie dachten, die Athena-Software sei für Ihre Forschung nützlich?
Alexandra Lubin: Als ich mit diesem Wirkstoffscreening begann, wussten wir, dass wir Automatisierung einführen mussten, da es nicht möglich sein würde, Tausende von Verbindungen manuell zu screenen. Als wir anfingen, uns verschiedene Methoden anzusehen, wurde deutlich, wie einfach das System zu bedienen und wie flexibel es war. Darüber hinaus wurden wir von der Tatsache angezogen Athena ermöglichte es uns, uns zu verzweigen und es bei Bedarf zu ändern.
Wo sehen Sie Ihre Arbeit als nächstes und die Auswirkungen?
Beth Payne: Ich glaube, dass es sicherlich die Tür geöffnet hat, um immer mehr Bildschirme aufzuführen. Vielleicht können wir jetzt ehrgeizigere Arten von Screens durchführen, bei denen wir beispielsweise mehr als einen Fluorophor oder mehr als einen Genotyp gleichzeitig verwenden, weil die Screening-Zeit so viel kürzer und effizienter ist, als Sie es können weitere Variablen einführen.
Über Dr. Elspeth Payne
Dr. Elspeth Payne ist leitende klinische Forscherin/klinische Beraterin am UCL Cancer Institute. Ihr Labor am Cancer Institute der UCL widmet sich der Untersuchung von erblichen Knochenmarkinsuffizienzerkrankungen und Leukämien und verwendet Zebrafische, um diese Krankheiten zu modellieren. Dr. Payne ist auch klinische Hämatologin am UCL-Krankenhaus, wo sie Menschen mit Bluterkrankungen wie Leukämie und Knochenmarkversagen behandelt.
![]() Über Dr. Alexandra Lubin
Über Dr. Alexandra Lubin
Dr. Lubin ist Postdoktorandin am UCL Cancer Institute, wo sie Zebrafische zur Untersuchung des myelodysplastischen Syndroms (MDS) und der akuten myeloischen Leukämie (AML) verwendet, um neuartige therapeutische Behandlungen zu entwickeln. Zuvor promovierte sie in Chemischer Biologie am Imperial College London, nachdem sie Chemie an der University of Cambridge studiert hatte.
Über IDEA Bio-Medical Ltd.

IDEA Bio-Medical konzentriert sich derzeit darauf, Zebrafischforscher zu befähigen, ihnen eine zuverlässige, robuste Lösung für die automatisierte und unvoreingenommene Bildanalyse von Zebrafischen bereitzustellen, indem das Wissen und die Expertise des Unternehmens angewendet werden.
Zu diesem Zweck entwickelte IDEA eine neuartige Deep-Learning-basierte Bildanalysesoftware für In-vivo-Zebrafischexperimente. Die Software erkennt automatisch die Zebrafischkontur und ihre inneren Organe im Hellfeld, ohne dass eine Benutzereingabe erforderlich ist. Die identifizierte Anatomie wird mit Fluoreszenzkanälen gekoppelt, um eine anatomiespezifische Untersuchung von Fluoreszenzänderungen zu ermöglichen. Es ist ein erschwingliches, benutzerfreundliches System, das speziell für die zuverlässige, automatisierte bildbasierte Analyse von Zebrafischen entwickelt wurde.
Die Software ist als eigenständiges Produkt erhältlich und akzeptiert Mikroskopiebilder in mehreren Bildformaten, einschließlich proprietärer. Es eignet sich für Forscher, die nur eine Handvoll Fische pro Woche abbilden und analysieren, sowie für Forscher, die Hunderte und Tausende von Fischen in Multi-Well-Platten für groß angelegte Bildschirme abbilden. IDEA Bio-Medical bietet ein neuartiges Pay-per-Use-Modell für den Zugriff auf die Software an, um einen flexiblen Zugriff zu ermöglichen. Daher können alle Forscher, die manuelle Mikroskope oder automatisierte Systeme anderer Anbieter verwenden, die Zfish-Software von IDEA problemlos verwenden, um bei Bedarf quantitative, aussagekräftige Informationen aus ihren Zebrafischbildern zu extrahieren.
Wenden Sie sich auf unserer Website an IDEA Bio-Medical Kontakt Formularund weitere Informationen finden Sie in der Zebrafisch-Analysesoftware Produktseite.
Richtlinie für gesponserte Inhalte: News-Medical.net veröffentlicht Artikel und verwandte Inhalte, die aus Quellen stammen können, mit denen wir bestehende Geschäftsbeziehungen unterhalten, vorausgesetzt, dass diese Inhalte einen Mehrwert für das zentrale redaktionelle Ethos von News-Medical.Net darstellen, das darin besteht, an Medizin interessierte Website-Besucher aufzuklären und zu informieren Forschung, Wissenschaft, medizinische Geräte und Behandlungen.
.

